Aufklärung der Strukturänderungen beim „Anschalten“ fluoreszierender Proteine
Kombination aus Messungen an Freie-Elektronen-Laser und Spektroskopie bringt neue Erkenntnisse
„Schaltbare“ fluoreszierende Proteine werden häufig als molekulare Marker in der hochauflösenden Lichtmikroskopie verwendet. Dadurch können Strukturen von wenigen Nanometern, d.h. milliardstel Meter Größe in lebenden Zellen sichtbar gemacht werden. Die verwendeten Proteine können durch Bestrahlung mit Licht einer bestimmten Wellenlängen reversibel zwischen einem nicht fluoreszierenden (Aus-) Zustand und einem fluoreszierenden (Ein-) Zustand hin und her geschaltet werden. Das Umschalten geschieht über viele ultraschnell ablaufende Prozesse und Zwischenzustände, die erst kürzlich strukturell charakterisiert wurden. Wie langsamer ablaufende Konformationsänderungen aussehen, war bisher unbekannt. Dies erschwert die umfassende Beschreibung des Schaltmechanismus auf molekularer Ebene.
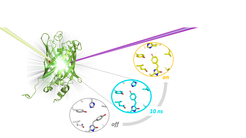
Forscher*innen der Abteilung für Biomolekulare Mechanismen am Max-Planck-Institut für medizinische Forschung in Heidelberg und Kollaborationspartner in Frankreich konnten nun erste Einblicke in den Mechanismus des fluoreszierenden Proteins rsEGFP2 geben. Für die Aufnahme der strukturellen Veränderungen dieses Biomarkers war die Kombination zweier Methoden ausschlaggebend: zeitaufgelöste serielle Röntgenkristallografie am Freien-Elektronen-Laser SACLA, Japan und transiente Absorptionsspektroskopie. Die Wissenschaftler haben dadurch die dreidimensionale Struktur eines entscheidenden Zwischenzustands bestimmt, der 10 Nanosekunden nach Bestrahlung des Proteins mit Licht entsteht. Die Ergebnisse zeigen die Reihenfolge der Ereignisse während des An- und Ausschaltens fluoreszierender Proteine. Dieses Verständnis kann zu einem verbesserten Design existierender und zukünftig genutzter schaltbare fluoreszierender Proteine für die Anwendungen in der hochauflösenden Lichtmikroskopie lebender Zellen beitragen.
